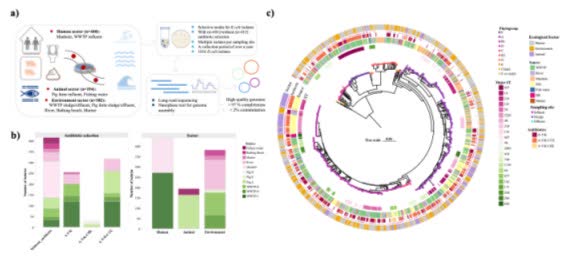
香港文匯報訊(記者 莫楠)香港大學工程學院團隊利用全港基因數據,研發一項嶄新的基因組追蹤方法,針對本港河流及污水等城市水體中常見、可產生抗生素耐藥性的大腸桿菌展開研究。團隊採用納米孔長讀長測序技術,分析一年內收集的1,016個大腸桿菌樣本,涵蓋不同細菌類型、耐藥基因及環狀質粒,以精準追蹤耐藥細菌和耐藥基因在本港不同環境的流動和傳播途徑。
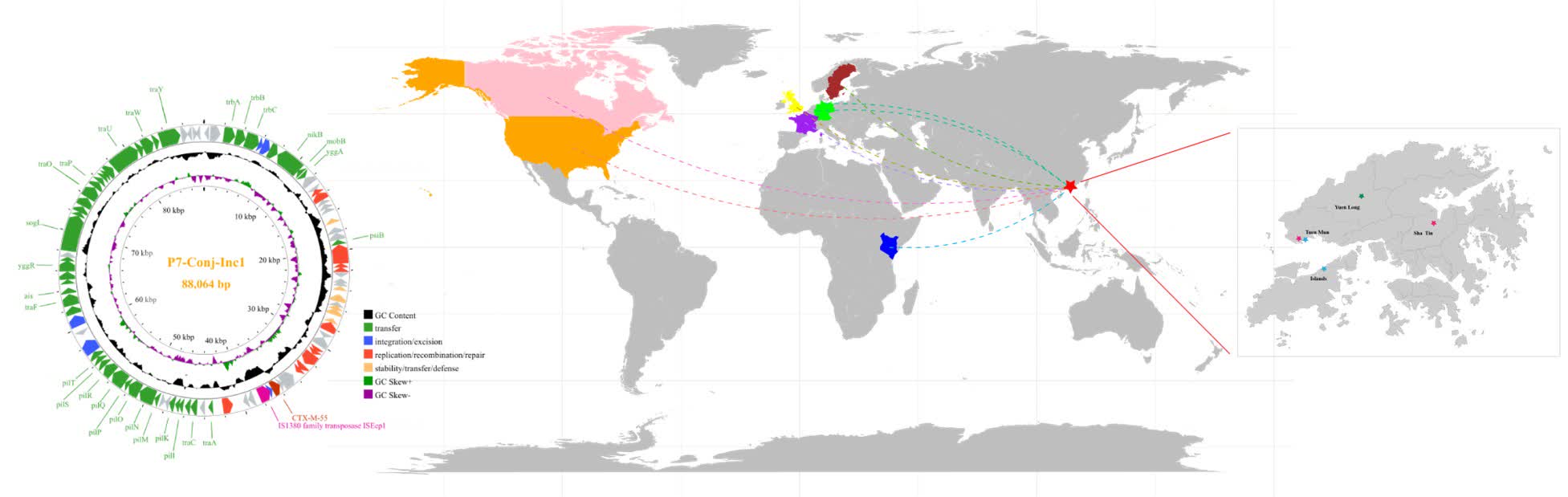
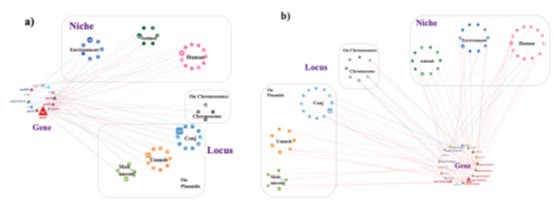
研究發現,不同來源的細菌在基因上高度相似,其中142組相同菌株同時存在於人體與環境水體中。團隊更識別出195個同時存在於人類、動物及環境的質粒,顯示耐藥基因能透過可移動DNA傳播。實驗室實驗證實,部分質粒可在細菌間轉移,為跨界傳播提供了實證支持,為公共衛生防護策略提供關鍵啟示。
這項研究由港大土木工程系教授張彤領導。團隊運用納米孔長讀長測序技術,對採集的樣本進行全面分析,從而實現全港高解析度的比對。研究將複雜基因數據轉化為實用工具,建立創新量化框架,用以測量細菌與耐藥基因在不同環境間的連通性。簡言之,城市水體已成為細菌與耐藥基因在人、動物與環境之間混合傳播的交匯點。
團隊表示發現對公共衛生至關重要,因當環境相互連通時,耐藥性將更快在人與環境間雙向傳播。研究第一作者徐曉慶表示:「微生物耐藥不僅關乎基因分佈的位置,更關乎它們在彼此連通的環境之間如何移動。透過量化人類、動物與環境水體之間的生態連通性,這項研究有助解釋耐藥性傳播途徑,並為更一體化的監測與干預策略提供依據。」
研究支持建立綜合監測系統,整合污水、環境及臨床數據,協助決策者及早預警並優先處理高風險質粒與菌株。此方法亦適用於其他城市,有助建立標準化基因組監測框架,在「同一健康」理念下評估及防控抗生素耐藥風險。
本研究獲張彤主持的大學教育資助委員會主題研究計劃資助,該計劃旨在支持本地資助大學圍繞戰略重點主題開展高水平科研。研究成果已發表於國際權威期刊《自然-通訊》。







評論(0)
0 / 255